Conceito
Primeiramente, é necessário definir a Biotecnologia para que, depois, seja apresentada a forma pela qual a Programação Computacional contribui para esse campo de conhecimento. Sendo assim, define-se biotecnologia como uma atividade fundamentada em princípios multidisciplinares, cujo objetivo é obter produtos úteis ou resolver problemas com o uso de agentes biológicos. Tais agentes biológicos podem abranger qualquer organismo vivo ou mesmo partes dele.
Tendo em vista esse conceito, apresenta-se a programação para biotecnologia como ferramenta para análise e processamento de dados biológicos, visando a utilização dos resultados como base para o desenvolvimento da área. Por meio da programação, é possível criar programas, algoritmos e modelos matemáticos que auxiliem profissionais a extrair conhecimento útil dos dados adquiridos, que podem ser muitos, principalmente quando o foco é a biologia.
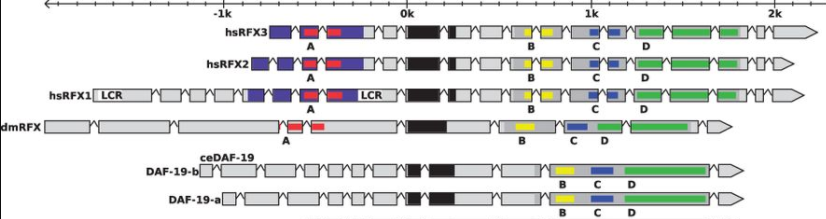
A programação para biotecnologia é capaz de criar programas e ferramentas para diversas finalidades. Pode-se citar, por exemplo, programas capazes de transcrever sequências de DNA (figura 1), de padronizar e automatizar protocolos de experimentos biológicos (figura 2), de criar imagens estruturais de genes (figura 3) e outras possibilidades.
Figura 1 - Programa do módulo BioPerl capaz de transcrever sequências de DNA
Figura 2 - Visão geral do programa Biocoder
Figura 3 - Imagem estrutural da família de genes RFX (presente em diversas espécies) gerada automaticamente pelo módulo do BioPerl chamado FeatureStack
Softwares
Como a biotecnologia está muito relacionada com a biologia molecular, muitos dos softwares, bancos de dados e linguagens utilizados nessa, também são recomendados para aquela. Dentre as linguagens mais utilizadas estão Python, Perl, Java, C, C++ e C#, com destaque para as duas primeiras (figura 4).
Figura 4- Logotipos das linguagens Perl e Python
Visando facilitar o trabalho com essas linguagens, muitos programadores e cientistas se reuniram para criar módulos e bibliotecas de código aberto, específicos para o uso e processamento de dados biológicos, com a finalidade principal de resolver problemas casuais. BioPython, BioPerl, BioJava e BioC++ são alguns desses módulos.
O uso de banco de dados também é essencial para essa área de conhecimento, já que dados biológicos são extensos. Sendo assim, é recomendado para os profissionais envolvidos nessa área que tenham conhecimento básico sobre bancos de dados, já que farão uso constante dos mesmos. São três tipos principais de bancos de dados biológicos: aqueles de arquivos simples, formado por uma coleção ordenada de arquivos de texto, os relacionais, que possuem as informações armazenadas em um conjunto de tabelas e os bancos orientados a objetos, que são sistemas de gerenciamento de dados associados à programação orientada a objetos, sendo robustos, capazes de administrar interações simultâneas de clientes e administrar objetos complexos. Os bancos ainda se dividem de acordo com o nível de manipulação de dados armazenados, podendo ser primários, possuindo informações biológicas originais, secundários, possuindo resultados de análises feitas a partir de dados primários e especializados, que armazenam dados de interesse específico.
Para trabalhar com biotecnologia, pode-se criar um banco de dados próprio, porém, o mais comum é utilizar bancos online, os quais disponibilizam dados para download e também dão a opção de fazer o upload de dados, quando o usuário tem interesse em compartilhá-los. Dentre os mais famosos estão:
- GenBank (primário): banco de dados criado pela famosa NCBI (National Center for Biotechnology Information) para sequências de de ácido nucléico.
- Protein Data Bank (primário): banco de dados de estruturas de proteínas.
- SWISS-Prot (secundário): banco de dados de sequência de proteínas.
- Flybase, HIV sequence database e Ribosomal Database Project (especializados): focados em organismos e informações específicos.
Os softwares também são usados como ferramentas na área da biotecnologia, facilitando a execução de pesquisas para aqueles com pouco conhecimento na área computacional. São diversas as ferramentas disponíveis para a pesquisa em Biotecnologia (tabela 1) , assim como suas funcionalidades que, em resumo, possibilitam: pesquisas biológicas na WEB, análise de sequências, alinhamento par-a-par, alinhamento de sequências, árvores e perfis, visualização de estruturas protéicas, predição de estrutura e função protéicas e pesquisa em genômica e proteômica.
Tabela 1: Softwares/Ferramentas disponíveis para a pesquisa na área de Biotecnologia
Código
<syntaxhighlight lang="python3">
- !/usr/bin/perl -w
- Procurar sequência de nucleotídeos em bloco de dados de sequências
my $alvo = "TAGCTG"; my $sequência_pesquisa =
'ACCTGACCCTGACCAGTTGACCCAATGTCAACTTCGGCTACAGGATTGACCCTGACCAAT'.
'CATCCTAAAGTGACTAGTCAGTAATTGGCATAGTCAGTACCCATGACTGATGGAGTAAAC'.
'ATGACCTGGATTAGCTGATATTTGCATGATCTAGCTAGACTGATCAGTACCCTAGATCCA'.
my @correspondências;
- Tentar encontrar uma correspondência nas letras 1-6 de $sequência_pesquisa e, em seguia, olhas as letras 2-7, # e assim por diante. Registrar o deslocamento inicial de cada correspondência.
foreach my $i (0..length $sequência_pesquisa) {
if ($alvo eq substr( $sequência_pesquisa, $i, length $alvo)){
push @correspondências, $i;
}
}
- Transformar @correspondências em uma lista separada por vírgulas para impressão print "Minhas correspondências ocorreram nos seguintes deslocamentos:
@correspondências. \n";
print "pronto\n";
</syntaxhighlight>
Por meio de demonstração de variáveis ($alvo, $sequência_pesquisa, @correspondências e $i) e loops, este programa é capaz de procurar uma sequência-alvo "TAGCTG" em uma sequência de dados e registrar a localização do início de cada correspondência.
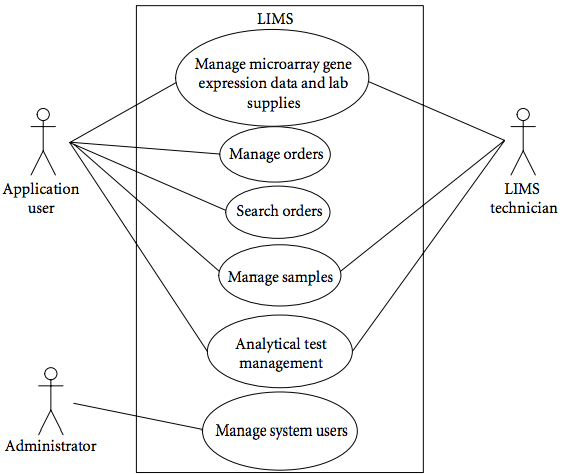
Diagrama de Casos de Uso
Figura 5 - O diagrama mostra alguns dos atores e serviços do MYCO-LIMS, um sistema de WEB capaz de oferecer serviços como gerenciamento de dados de expressão genética e de materiais laboratoriais, além de outras funcionalidades não ligadas à biotecnologia.
Interações com as tecnologias
Conceito de Mineração de Dados: Uma vez que os conjuntos de dados biológicos utilizados na Programação para Biotecnologia são considerados grandes, é necessário o uso da mineração de dados, que é um processo que possibilita a localização, a interpretação e a avaliação de padrões em grandes conjuntos de dados.
Tecnologia Banco de Dados: Como destacado nos tópicos anteriores, a Programação para biotecnologia lida com dados biológicos, os quais tem como característica a a existência em grande quantidade. Dessa forma, o uso de banco de dados para guardar e administrar dados é recomendável.
[1]: Nanotecnologia: a criação de sistemas capazes de desenhar nanosensores que detectam pequenas concentrações de certas substâncias químicas é só uma das possibilidades criadas pelo avanço na Programação para Biotecnologia.
[2]: CUDA: é uma plataforma de computação paralela que possibilita aumentos consideráveis na performance computacional, causados pelo aproveitamento da potência da unidade de processamento gráfico (GPU). Na Programação, por exemplo, o CUDA permite o envio de códigos em C, C++ e Fortran diretamente à GPU, não havendo necessidade de usar uma linguagem de compilação. Resumindo, é possível migrar partes de aplicativos com alta demanda computacional para a GPU utilizando o CUDA Toolkit. Isso possibilita a melhoria de processos que lidam com simulações de alta demanda, algo comum na Biotecnologia. Por exemplo, a simulação molecular NAMD (dinâmica molecular em nanoescala), relacionada à Biotecnologia, alcança um grande aumento de performance.
Informações adicionais
Riscos biotecnológicos
Nem tudo é previsível no ramo da Biotecnologia, ainda que seu objetivo seja criar soluções para problemas. Alguns riscos estão inerentes à manipulação e movimentação de genes, como a perda de ecossistemas existentes e a criação e liberação de patógenos altamente destrutivos.
As pesquisas de adição de novas funções em organismos, mesmo que realizadas com o objetivo de evitar problemas, podem resultar na liberação de patógenos mais perigosos do que aqueles que ocorrem naturalmente e incentivar modificações perigosas por parte de outros pesquisadores.
A tecnologia e a compreensão de manipulação de genes e organismos utilizadas na Biotecnologia estão cada vez mais baratas e mais acessíveis, o que aumenta ainda mais os riscos de liberação acidental. A menos que as contramedidas sejam desenvolvidas, um cenário mais perigoso pode ocorrer se pessoas com acesso a essa tecnologia e entendimento comecem a colaborar com grupos de ideologia radical dispostos a usar medidas extremas para atingir seus objetivos.
Referências bibliográficas
Profissões do Futuro: bioinformática transforma dados em conhecimento. Disponível em: <http://educacao.estadao.com.br/noticias/geral,bioinformatica-transforma-dados-em-conhecimento,1716455>. Acesso em: 22 set. 2017.
BIOINFORMÁTICA – O LADO DO PROGRAMADOR. Disponível em: <https://www.revista-programar.info/artigos/bioinformatica-o-lado-do-programador/ https://futureoflife.org/background/risk-of-biotechnology/>. Acesso em: 22 set. 2017.
LGE - Ferramentas. Disponível em: <http://www.lge.ibi.unicamp.br/pagina/estrutura/montagem_script/exibe_ferramenta_pagina.php>. Acesso em: 30 set. 2017.
XIONG, J. Essential Bioinformatics. Cambridge University Press ; ISBN: 0-521-60082-0; 339pp., 2006.
MALAJOVICH M. A. Biotecnologia. Segunda Edição, atualizada. Rio de Janeiro, 2016.
GIBAS, C.; JAMBECK, P. Desenvolvendo Bioinformática: ferramentas de software para aplicações em biologia. Rio de Janeiro: Editora Campus, 2001.
BENEFITS & RISKS OF BIOTECHNOLOGY. Disponível em: <https://futureoflife.org/background/risk-of-biotechnology/>. Acesso em: 30 set. 2017.
ANANTHANARAYANAN, V.; THIES, W. Biocoder: A programming language for standardizing and automating biology protocols. Journal of Biological Engineering, Volume 4, 13p., 2010.
FRECH, C.; CHOO, C.; CHEN, N. FeatureStack: Perl module for comparative visualization of gene features. Bioinformatics, Volume 28, Seção 23, páginas 3137–3138, 2012.